Autor: dipl. biolog – master Vladimir Vukić
E-mail: vladimirvukic@gmail.com
.
PCR se može kombinovati sa drugim tehnikama i time analizu učiniti još specifičnijom. Umnoženi fragmenti se mogu tretirati endonukleazama (PCR-RFLP), sekvencionirati, analizirati razlike u konformaciji jednolančanih DNK molekula (PCR/SSCP) ili istovremeno umnožiti dva ili više fragmenata sa različitim prajmerima (multiplex PCR). Takođe se mogu koristiti i tehnike kao što je analiza slučajno umnoženih polimorfnih DNK (RAPD), pri kojoj se koriste kratki, jednosmerni i slučajni prajmeri, analiza mikrosatelita (SSR) ili analiza polimorfnosti dužine umnoženih fragmenata DNK (AFLP).
Jedan od ključnih aspekata pri analizi hrane je i kvantifikacija, naročito kod alergena. Kvantifikacija se vrši putem kvantitativno-kompetitivne PCR (QC-PCR) i real-time PCR (RT-PCR). Upotreba specifičnih proba ili obeleženih prajmera omogućava da se prati tok reakcije i kvantifikuje krajnji produkt.
.
PCR/RFLP – Restriction Fragment Lenght Polymorphism
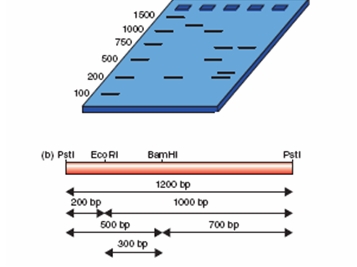
Nakon PCR-a umnožena DNK se tretira restrikcionim enzimima (endonukleazama). Dobijeni fragmenti se razdvajaju elektroforezom. Princip metode je da u zavisnosti od prisutnosti resrtikcionih mesta dobijamo DNK fragmente različite dužine (Sl. 2). Poređenjem dva profila može se utvrditi da li su DNK molekuli izolovani iz iste ili različitih vrsta.
.
PCR/SSCP – Single-strand Conformational Polymorphism
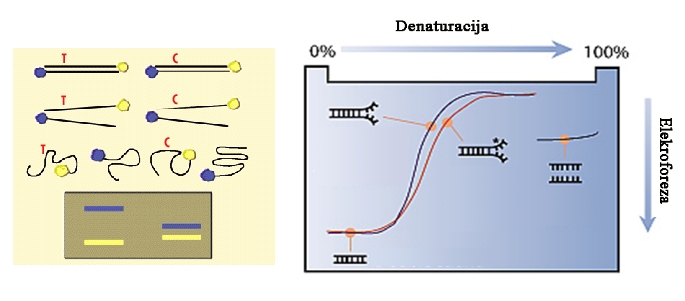
Metoda se zasniva na poređenju pređenog elektroforetskog puta jednolančanih DNK (Sl. 3). Dobijeni PCR produkti se denaturišu zagrevanjem i razdvoje elektroforetski. Usled razlika u DNK sekvenci, različiti molekuli će imati različiti trodimenzionalni oblik, što će usloviti da se lakše ili teže kreću kroz gel, odnosno pređu različiti put. Prajmere treba odabrati tako da DNK izolovana iz iste vrste pređe isti put, odnosno da budu specifični za vrstu.
.
Sekvencioniranje
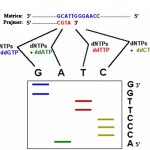
Jedna od mogućih metoda analize nakon PCR umnožavanja je sekvencioniranje produkata Sangerov-ov metodom (Sl 4). Naziva se još i dideoksi sekvencioniranje jer se bazira na prisustvu dideoksinukleozid trifosfata (ddNTP), koji se od običnih deoksinukleozid trifosfata razlikuju po nedostatku 3’ hidroksilne grupe. Metoda se bazira na in vitro sintezi DNK. Kao matrica se koristi DNK čija je sekvenca nepoznata. Poznati prajmer je radioaktivno obeležen. Nezavisno se rade četiri reakcije uz dodatak DNK polimeraze i uobičajenih dNTP, a u svaku se dodaje određeni ddNTP. Princip je da svaki put kada polimeraza ugradi ddNTP prekida dalju sintezu.
Razvojem nauke i tehnologije dati metod je usavršavan. Radioaktivni obeleživači su zamenjeni fluorescentnim bojama, a reakcija se odvija u sekvencioneru, aparatu koji sve radi automatski od unošenja uzoraka do čitanja rezultata sa gela. Dalji napredak je bio uvođenje četiri različite boje za četiri reakcije, što je omogućilo da se elektroforetsko razdvajanje obavlja na jednom gel sistemu, i na kraju obeležavanje samih ddNTP fluorescentnim bojama, čime je omogućeno da se sve odvija u jednoj reakcionoj smeši i razdvaja na jednom gelu.
.
Multiplex PCR
U jednoj reakcionoj smeši se koristi više prajmera, što omogućuje istovremeno umnožavanje više željenih fragmenata. Na taj način se može istovremeno ispitati prisustvo ili odsustvo više mikroorganizama u prehrambenim proizvodima, ili detektovati tačan kvalitativni sastav pojedinih proizvoda koji su smeša sasatojaka različitog porekla.
.
AFLP – Amplified fragment lenght polymorphism
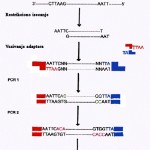
AFLP metoda kombinuje restrikciono isecanje i PCR (Sl. 5). Prvi korak je da se DNK iseče sa dva tipa restrikcionih enzima: jedan koji prekida lanac češće (prepoznaje restrikciona mesta od 4 bazna para) i drugi koji prekida lanac ređe (prepoznaje restrikciona mesta od 6 baznih parova). Na dobijene fragmente se dodaju specifični adapteri. Prajmeri koji se koriste za umnožavanje imaju komplementarnu sekvencu adapteru i nasumično dodata jedan, dva ili tri nukleotida (A, T, C ili G). Ovo omugućuje specifičnost prajmera i umnožavanje samo jednog dela fragmenata DNK, na osnovu čega se može utvrditi da li ispitivani uzorak pripada datoj vrsti. PCR umnožavanje se obično izvodi u dva ciklusa. U prvom se adapterima dodaje jedan nukleotid, što dovodi do umnožavanja samo dela fragmenata, a u drugom se dodaju još jedan ili dva, što dovodi do umnožavanja samo dela prethodno umnoženih fragmenata, te povećava specifičnost. AFLP je veoma robustna metoda što je čini pogodnom za upotrebu pri testovima identifikacije genotipova.
.
Real-time PCR
Real-time PCR omogućuje kvantifikaciju umnoženog segmenta DNK, a time i zastupljenost u uzorku. Real-time PCR se razlikuje od obične PCR po tome što ima detektor fluorescencije.
Real Time PCR test omogućuje detekciju i kvantifikaciju umnoženog segmenta DNK u realnom vremenu, u toku amplifikacije uzorka. Postoje tri varijacije ove procedure u smislu finalne detekcije količine PCR proizvoda amplifikacije:
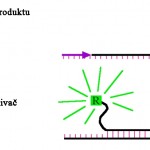
1. Molekularni svetionik – jednolančani segment DNK koji stvara ukosnicu čija se petlja sastoji od baza koje su komplementarne lancu željenog PCR produkta. Krajevi ove strukture sadrže fluorescentnu reporter (R) boju i prigušivač (Q) boju. R ekscitira odgovarajućim svetlosnim signalom, a emitovanu svetlost apsorbuje Q, pa se ne detektuje fluorescencija. Tokom PCR-a novosintetisani lanci se na visokoj temperaturi denaturišu, te se narušava i struktura ukosnice. Pri snižavanju temperature radi vezivanja prajmera, svetionik se komplementarno vezuje sa nekim PCR produktima (Sl. 6). Ukoliko se PCR produkt vezao, onemogućeno je Q elementu da priguši fluorescenciju i emitovana svetlost se detekuje detektorom fluorescencije.
2. TaqMan proba ima dva tipa fluorofora (Q i R) koje se nalaze na krajevima ove sekvence (Q na 3’ a R na 5’ kraju). Q emituje svetlost velike talasne dužine (crvene boje) i time prikriva signal koji emituje R (mala talasna dužina, zelene boje). To se postiže putem transfer fluorescentne rezonantne energije koja predstavlja inhibiciju jedne boje drugom bez emisije protona. Proba se vezuje za ciljnu DNK, nakon čega Taq polimeraza sintetiše komplementaran lanac počevši od prajmera uklanjajući vezanu TaqMan probu koja joj se nađe na putu. Tako se odvajaju i R i Q pa se sa R emituje energija koja se kvantifikuje (Sl. 7).
3. SYBR Green metoda se prva koristila za Real Time PCR, vezuje se za DNK i emituje zelenu svetlost u pobuđenom stanju (pobuđuje se plavom svetlošću).
Nivo emisije fluorescencije je proporcionalan zastupljenosti ciljnih sekvenci u uzorku. Koncentracija DNK je proporcionalna broju ciklusa u toku eksponencijalne faze PCR reakcije. Svaka analiza sadržaja ciljne sekvence u uzorku uključuje i seriju standarda, pa se na osnovu toga može odrediti njen udeo pomoću standardne krive.
.
QC/PCR (quantitative competitive PCR)
Kod kvantitativno kompetitivne PCR procedure količina ciljne sekvence u uzorku se određuje poređenjem količine umnožene ciljne DNK sa kompetitivnim standardom. Koncentracija kompetitorne DNK koja se dodaje je poznata i ona koamplificira sa DNK koja se ispituje. Prajmer se vezuje i za DNK kompetitora, ali na drugim mestima, pa se dobija fragment različite veličine u odnosu na ciljni uzorak, što omugućuje razdvajanje na gelu. Ukoliko je intenzitet trake PCR proizvoda isti onda je početna količina DNK jednaka količini kompetitorne DNK.
.
Literatura
1. Anon. (1999): Microbiology of food and feeding stuffs— Protocol for the validation of alternative methods. Document established by the Joint Group of MicroVal (WG 6) and CEN (WG 6/TAG 2). ISO/DIS 16140, Geneva, Switzerland.
2. Anon. (1999A): Microbiology of food and animal feeding stuff—carcass sampling for microbiological analysis. ISO/CD 17604. International Organization for Standardization, Geneva, Switzerland.
3. Anon. (2000): Microbiology of food and animal feeding stuff—Preparation of test samples, initial suspension and decimal dilution for microbiological examination. Doc CEN/TC 275/ WG 6 N 114. Published by AFNORE, Paris, France.
4. Anon. (2001): Protocol for the validation of alternative microbiological methods. NV-DOC.D-2001-04-25. Published by NordVal. Copenhagen, Denmark.
5. Arlorio, M., Coïsson, J. D., Cereti, E., Travaglia, F., Capasso, M., Martelli, A. (2003): Eur Food Res Technol 216: 253–258.
6. Asensio, L., González, I., Fernández, A., Céspedes, A., Hernández, P. E., García, T., Martín, R. (2000): J Food Prot 63: 1248–1252.
7. Asensio, L., González, I., Fernández, A., Rodríguez, M. A., Hernández, P. E., García, T., Martín, R. (2001): J Agric Food Chem 49: 1720–1723.
8. Asensio, L., González, I., Fernández, A., Rodríguez, M. A., Lobo, E., Hernández, P. E., García, T., Martín, R. (2001): Arch Lebensmittelhyg 52: 131–134.
9. Asensio, L., González, I., Fernández, A., Rodríguez, M. A., Lobo, E., Hernández, P. E., García, T., Martín, R. (2002): J Food Prot 65: 432–435.
10. Bania J, Ugorski M, Polanowski A, Adamczyk E (2001) J Dairy Res 68:333–336
11. Borst, A., Box, A. T., Fluit A. C. (2004): False-positive results and contamination in nucleic-acid amplification assays: Suggestions for a “prevent and destroy“ strategy. Eur. J. Clin. Microbiol. Infect. Dis. 23: 289-299.
12. Brezná, B., Hudecová, L., Kuchta, T. (2006): Eur Food Res Technol 223: 373–377.
13. Busconi, M., Foroni, C., Corradi, M., Bongiorni, C., Cattapan, F., Fogher, C. (2003): Food Chem 83: 127–134.
14. Céspedes, A., García, T., Carrera, E., González, I., Hernández, P. E., Martín, R. (1999): J Agric Food Chem 47: 1046–1050.
15. Doveri, S., O’Sullivan, D. M., Lee, D. (2006): J Agric Food Chem 54: 9221–9226.
16. Hernández, M., Rodríguez-Lazaro, D., Zhang, D., Esteve, T., Pla, M., Prat, S. (2005): J Agric Food Chem 53: 3333–3337.
17. Holst-Jensen, A., Rønning, S. B., Løvseth, A., Berdal, K. G. (2003): Anal Bioanal Chem 375: 985–993.
18. Holzhauser, T., Wangorsch, A., Vieths, S. (2000): Eur Food Res Technol 211: 360–365.
19. Köppel, E., Stadler, M., Lüthy, J., Hübner, P. (1998): Z Lebensm Unters Forsch A 206: 399–403.
20. Leimanis, S., Hernández, M., Fernández, S., Boyer, F., Burns, M., Bruderer, S., Glouden, T., Harris, N., Kaeppeli, O., Philipp, P., Pla, M., Puigdomènech, P., Vaitilingom, M., Bertheau, Y., Remacle, J. (2006): Plant Mol Biol 61: 123–139.
21. López-Calleja, I., Alonso, I. G., Fajardo, V., Rodríguez, M. A., Hernández, P. E., García, T., Martín, R. (2005): Int Dairy J 15: 1122–1129.
22. López-Calleja, I., González, I., Fajardo, V., Martín, I., Hernández, P. E., García, T., Martín, R. (2005): J Dairy Sci 88: 3115–3120.
23. López-Calleja, I., González, I., Fajardo, V., Martín, I., Hernández, P. E., García, T., Martín, R. (2007): Int Dairy J 17: 729–736.
24. Lopparelli, R. M., Cardazzo, B., Balzan, S., Giaccone, V., Novelli, E. (2007): J Agric Food Chem 55: 3429–3434.
25. Macrina, F.L. (1995): Scientific Integrity: An Introductory Text with Ceses. ASM Press, Washington, DC.
26. Mafra, I., Ferreira, I. M. P. L. V. O., Beatriz, M., Oliveira, P.P. (2007): Food authentication by PCR-based methods. Universidado de Porto, Porto.
27. Mafra, I., Ferreira, I. M. P. L. V. O., Faria, M. A., Oliveira, B. P. P. (2004): J Agric Food Chem 52: 4943–4947.
28. Matsunaga, T., Chikuni, K., Tanabe, R., Muroya, S., Shibata, K., Yamada, J., Shinmura, Y. (1999): Meat Sci 51: 143–148.
29. Maudet, C., Taberlet, P. (2001): J Dairy Res 68: 229–235.
30. Meyer, R., Höfelein, C., Lüthy, J., Candrian, U. (1995): J AOAC Int 78:1542–1551.
31. Montiel-Sosa, J. F., Ruiz-Pesini, E., Montoya, J., Roncalés, P., López-Pérez, M. J., Pérez-Martos, A. (2000): J Agric Food Chem 48:2829–2832.
32. Pasqualone, A., Montemurro, C., Grinn-Gofron, A., Sonnante, G., Blanco, A. (2007): J Agric Food Chem 55: 3312–3318.
33. Regulation (EC) No 1829/2003 of the European Parliament and of the Council of 22 September 2003 on genetically modiWed food and feed. OV J Eur Union L 268:1–23
34. Regulation (EC) No 1830/2003 of the European Parliament and of the Council of 22 September 2003 concerning the traceability of food and feed products produced from genetically modiWed organisms and amending Directive 2001/18/EC. OV J Eur Union L 268:24–28
35. Russel, V. J., Pryde, S. E., Santos, A. T., Rosa, C., Quinteiro, J., Rey-Méndez, M. (2002): Eur Food Res Technol 214: 171–177.
36. Rønning, S. B., Rudi, K., Berdal, K. G., Holst-Jensen, A. (2005): J Agric Food Chem 53: 8874–8880.
37. Sanchez, S. (2006): Making PCR a normal routine of the food microbiology lab. The University of Georgia, Athens.
38. Stephan, O., Vieths, S. (2004): J Agric Food Chem 52: 3754–3760.
39. Wellinghausen. N., Wirths, B., Essig, A., Wassill, L. (2004): Evaluation of the Hyplex Bloodscreen multiplex PCR-enzyme-linked immunosorbent assay system for direct identification of gram-positive cocci and gram-negative bacilli from positive blood cultures. J. Clin.Microbiol. 43: 3147-3152.
40. Wolf, C., Lüthy, J. (2001): Meat Sci 57: 161–168.
41. Wolf, C., Rentsch, J. (1999): J Agric Food Chem 47: 1350–1355.
42.http://www.greenfacts.org/
43.http://www.celiac.org/
44.http://www.bio.davidson.edu/